Investigadores de la Facultad de Salud Pública Mailman de la Universidad de Columbia han desarrollado un novedoso método informático diseñado para identificar biomarcadores proteicos asociados a enfermedades complejas, entre ellas el Alzheimer.
Esta innovadora línea computacional, denominada MR-SPI (Mendelian Randomization by Selecting genetic instruments and Post-selection Inference) analiza los biomarcadores que pueden inducir cambios estructurales tridimensionales en las proteínas, lo que proporciona una visión clave de los mecanismos de la enfermedad y pone de relieve posibles dianas para la intervención terapéutica. Estos hallazgos, publicados en Cell Genomics, podrían conducir a avances en la detección precoz y las estrategias de tratamiento de la enfermedad de Alzheimer.
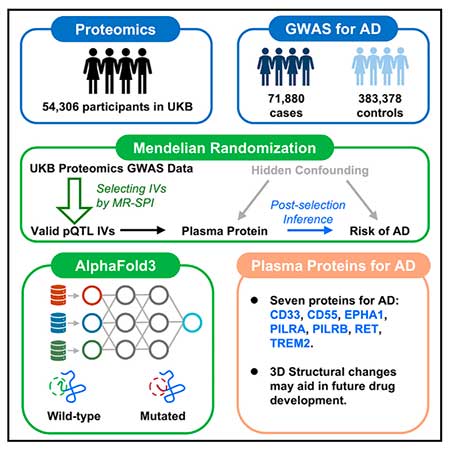
Esta metodología computacional desarrollada por la Columbia Mailman School revela posibles nuevas dianas terapéuticas para la Enfermedad de Alzheimer
Tal y como afirma afirma el investigador principal Zhonghua Liu, ScD, profesor adjunto de Bioestadística en la Columbia Mailman School, «la enfermedad de Alzheimer se define por la presencia en el cerebro de placas de beta-amiloide y ovillos neurofibrilares de tau, que se acumulan décadas antes de que aparezcan los síntomas. El diagnóstico precoz actual requiere muchos recursos o es invasivo. Además, las terapias actuales contra el Alzheimer dirigidas a la beta-amiloide proporcionan cierto alivio sintomático y pueden ralentizar la progresión de la enfermedad, pero no llegan a detenerla por completo».
«Nuestro estudio subraya la urgente necesidad de identificar biomarcadores proteínicos sanguíneos menos invasivos y más accesibles para la detección precoz de la enfermedad de Alzheimer. Tales avances podrían desentrañar los mecanismos subyacentes de la enfermedad y allanar el camino para tratamientos más eficaces», recalca el investigador.
Proteínas clave relacionadas con el riesgo de padecer Alzheimer
Utilizando datos del Biobanco del Reino Unido, que incluye a 54.306 participantes, y un estudio de asociación de genoma completo (GWAS) con 455.258 sujetos (71.880 casos de EA y 383.378 controles), el equipo de investigación identificó siete proteínas clave -TREM2, PILRB, PILRA, EPHA1, CD33, RET y CD55- que presentan alteraciones estructurales relacionadas con el riesgo de padecer Alzheimer.
«Descubrimos que algunos fármacos aprobados por la FDA que ya se dirigen a estas proteínas podrían reutilizarse para tratar el Alzheimer», indica Liu, destacando que «nuestros hallazgos subrayan el potencial de esta línea para identificar biomarcadores proteicos que puedan servir como nuevas dianas terapéuticas, así como para proporcionar oportunidades de reutilización de fármacos en la lucha contra el Alzheimer».
«La MR-SPI es especialmente valiosa para dilucidar relaciones causales en enfermedades complejas como el Alzheimer, en las que los métodos tradicionales tienen dificultades», recalca el profesor adjunto de Bioestadística en la Columbia Mailman School.
«La integración de MR-SPI con AlphaFold3 -una herramienta avanzada de predicción de estructuras tridimensionales de proteínas- mejora aún más su capacidad para predecir cambios estructurales tridimensionales causados por mutaciones genéticas, lo que proporciona una comprensión más profunda de los mecanismos moleculares que conducen a la enfermedad», afirma Liu.
Las personas interesadas pueden consultar aquí el artículo «Deciphering proteins in Alzheimer’s disease: A new Mendelian randomization method integrated with AlphaFold3 for 3D structure prediction» publicado en Cell Genomics.














